
Desenvolvimento de marcadores microssatélites para análises de diversidade genética em espécies de Philodendron (Araceae)
Rômulo da Fonsêca dos Santos*
![]() https://doi.org/10.63753/osaber.a15n15.62
https://doi.org/10.63753/osaber.a15n15.62
RESUMO
Philodendron é um dos gêneros neotropicais mais importantes da família Araceae, tanto pela sua representatividade em número de espécies quanto pelo seu destaque na floricultura. Considerando que o êxito na conservação das espécies vegetais de importância ecológica e econômica depende da manutenção de sua diversidade genética e que esta informação não se encontra disponível para a maioria das espécies da família Araceae, o presente estudo teve como objetivo desenvolver marcadores moleculares microssatélites (SSR) para P. solimoesense e determinar o potencial de transferibilidade em P. goeldii, P. leal-costae e P. venezuelense. Para tal, foi construída uma biblioteca de SSR nucleares, obtida por meio de sequenciamento shotgun do DNA genômico de P. solimoesense em um sequenciador Roche 454 GSFLX. A ocorrência e as frequências dos SSRs foram determinadas utilizando-se o software SciRoKo. Um total de 2.299 loci de SSR foram encontrados, dos quais 1.168 contêm motivos SSR aptos para serem utilizados no desenho de primers, constituindo mais de 50% de todas as regiões observadas. Destas, as 28 regiões contendo os maiores números de repetições foram selecionadas para o desenvolvimento dos marcadores a partir da ferramenta WebSat. Os testes de amplificação dos 28 marcadores via PCR de gradiente usando diferentes temperaturas de anelamento, e a análise por eletroforese em gel de agarose 1,8% foram bem-sucedidos. Para análise de polimorfismo e transferibilidade, foram selecionados 8 marcadores que amplificaram na temperatura de 58 ºC (Psol-0197, Psol-1036, Psol-0781, Psol-0313, Psol-0909, Psol-0116, Psol-0101, Psol-0398). Todos os primers desenvolvidos para P. solimoesense amplificaram fragmentos em P. goeldii, demonstrando um percentual de 100% de transferibilidade. Observou-se um percentual de amplificação cruzada de 75% em P. venezuelense e 50% em P. leal-costae. Os resultados mostraram que os primers desenvolvidos para identificar locos de SSR polimórficos em populações de P. solimoesense apresentaram porcentagem considerável de amplificação cruzada.
Palavras-chave: SSR. Floresta Amazônica. Mata Atlântica. Diversidade genética.
ABSTRACT
Philodendron is one of the most important neotropical genera of the Araceae family, both for its representativeness in number of species and for its prominence in floriculture. Considering that the success in the conservation of plant species of ecological and economical importance depends on the maintenance of their genetic diversity and that this information is not available for most species in the Araceae family, the present study aimed to develop microsatellite molecular markers (SSR ) for P. solimoesense and determine the transferability potential in P. goeldii, P. leal-costae and P. venezuelense. To do so, a nuclear SSR library was produced, obtained through shotgun sequencing of P. solimoesense genomic DNA on a Roche 454 GSFLX sequencer. The occurrence and frequencies of SSRs were determined using the SciRoKo software. A total of 2,299 SSR loci were found, of which 1,168 contain SSR patterns suitable to be used in primer design, constituting more than 50% of all regions observed. From this number, the 28 regions containing the highest number of repetitions were selected for the development of markers using the WebSat tool. Amplification tests of the 28 markers via gradient PCR using different annealing temperatures, and analysis by electrophoresis in 1.8% agarose gel were successful. For polymorphism and transferability analysis, 8 markers that amplified at a temperature of 58 ºC were selected (Psol-0197, Psol-1036, Psol-0781, Psol-0313, Psol-0909, Psol-0116, Psol-0101, Psol-0398 ). All the primers developed for P. solimoesense amplified fragments in P. goeldii, demonstrating 100% transferability. A cross-amplification percentage of 75% was observed in P. venezuelense and 50% in P. leal-costae. The results showed that the primers developed to identify polymorphic SSR loci in P. solimoesense populations presented a considerable percentage of cross-amplification.
Keywords: SSR. Amazon Rainforest. Atlantic Forest. Genetic diversity.
INTRODUÇÃO
A partir da caracterização dos recursos genéticos de suas espécies nativas, o gerenciamento e a manutenção da diversidade de comunidades vegetais de florestas tropicais tornam-se possíveis. Neste âmbito, diversas investigações baseadas em técnicas de Biologia Molecular são indispensáveis para fins de conservação, permitindo a identificação de regiões com maiores níveis de diversidade ou áreas de isolamento (devido a processos de fragmentação), propiciando, por exemplo, o manejo de habitats, com o objetivo de minimizar ou reduzir a extinção de espécies (Pennington & Dick, 2010). Além disso, a caracterização da diversidade genética de espécies vegetais de centros de biodiversidade relevantes, como a bacia amazônica e a Floresta Atlântica, pode contribuir para o entendimento da ecologia, como também da evolução desses sensíveis ecossistemas, fornecendo informações valiosas para estudos populacionais e biogeográficos das regiões de interesse (Miyaki & ALves, 2006).
As florestas Amazônica e Atlântica são consideradas as florestas tropicais mais abundantes em elementos da fauna e da flora e compreendem duas das principais fontes de biodiversidade do planeta (Costa, 2003). Atualmente, estes dois biomas basicamente úmidos e sombreados encontram-se separados por uma extensa faixa de campos abertos de clima mais seco (Cerrado e Caatinga), porém há vários indícios de que, no passado, haviam conexões entre a Amazônia e a Floresta Atlântica, as quais se mostram em um padrão muito mais complexo do que se pensava anteriormente (Turchetto-Zolet et al., 2012; Batalha-Filho et al., 2013). Os padrões evolutivos entre as populações existentes nos dois biomas ainda não estão muito claros, especialmente no que diz respeito à ocupação e estruturação dos ambientes pelas comunidades vegetais.
Diversas técnicas de Biologia Molecular podem ser utilizadas na caracterização da distribuição espacial da variabilidade e conectividade genética entre populações vegetais (naturais ou cultivadas), quer sejam contínuas ou claramente fragmentadas (Weising et al., 2005). Dentre os marcadores mais utilizados nas análises populacionais, encontram-se os SSRs, ou microssatélites, que se destacam por serem facilmente acessados através de metodologias muito bem estabelecidas, além de serem extremamente informativos devido ao alto nível de polimorfismo acessado e por seu caráter codominante (Weising et al., 2005; Victoria et al., 2011). Contudo, o desenvolvimento e a aplicação de marcadores informativos para a imensa maioria das plantas nativas ainda são bastante limitados.
O gênero Philodendron apresenta uma grande diversidade ecológica (Mayo et al., 1997) e é o segundo maior da família Araceae, com aproximadamente 500 espécies conhecidas (The Plant List, 2013), sendo um dos principais componentes da flora da América tropical. As espécies que compõem o gênero possuem um potencial econômico significativo, levando em consideração sua utilização tradicional na alimentação, fibras para artesanato e, principalmente, no âmbito ornamental, por conta das folhas conspícuas e morfologia variada (Oliveira, 2011; Castelar et al., 2013).
Embora estejam sendo desenvolvidas algumas análises de sistemática molecular na tentativa de entender as complexas relações filogenéticas entre as espécies de Philodendron e os grupos irmãos (e.g. Gauthier et al., 2008), dados relacionados à análise da diversidade genética entre populações nativas das espécies do grupo são escassos, havendo uma completa carência da descrição de marcadores moleculares informativos para tais abordagens.
MATERIAL E MÉTODOS
Foram utilizados espécimes (uma folha jovem por indivíduo) de diferentes populações de espécies de Philodendron provenientes da Amazônia e da Floresta Atlântica. O procedimento de extração de DNA utilizado foi de acordo com o protocolo CTAB I descrito por Weising et al. (2005), com modificações. Aproximadamente 2g de folhas jovens foram lavadas e pulverizadas em um almofariz com nitrogênio líquido, utilizando um pistilo. Após a transferência do material para tubos de polipropileno de 50 ml, foram adicionados 10 ml de tampão de extração [CTAB 2%, Tris-HCl 0,1 mM (pH 8,0), EDTA 20 mM (pH 8,0), NaCl 1,4 M NaCl e β-mercaptoetanol 2%], a 60 °C, por pelo menos 40 min, sob leve agitação periódica. Após o resfriamento do extrato, foram adicionados 10 ml de solução de clorofórmio e álcool isoamílico (24:1) aos tubos, efetuando-se uma homogeneização seguida de centrifugação por 20 min a 4000 rpm. Parte do sobrenadante resultante (~8 ml) foi transferida para um novo tubo, adicionando-se 60% do volume de isopropanol, misturando-se cuidadosamente por inversão. Posteriormente, os tubos foram centrifugados a 4000 rpm por 20 min, ocorrendo, assim, a precipitação do DNA, e, então, o pellet foi lavado com etanol 70%.
A precipitação seletiva de polissacarídeos contaminantes foi realizada conforme descrito por Michaels e colaboradores (1994), com algumas modificações: (1) ao pellet de DNA foram adicionados 500 uL da solução de precipitação [Tris-HCl 10 mM (pH 8,0) e NaCl 250 mM]; (2) ao DNA diluído foram adicionados 180 uL de etanol absoluto gelado sob agitação e, então, submetidos a −20 °C por 18 h; (3) o material foi centrifugado a 14000 rpm por 20 min a 4 °C e a fase líquida (sobrenadante) foi transferida para um novo tubo; (4) foram adicionados 700 uL de isopropanol os tubos contendo o DNA diluído, os quais foram invertidos por 50 vezes e, então, deixados por 15 min a temperatura ambiente; (5) o material foi centrifugado a 14000 rpm por 15 min a 24 °C, e, então, o sobrenadante foi descartado para possibilitar a secagem do pellet a temperatura ambiente. O DNA obtido foi ressuspendido em 400 uL de solução TE [Tris-HCl 10 mM (pH 8,0) e EDTA 0,1 mM], e, então, foi adicionado 1 uL de RNase 10 ug/ml para a digestão do RNA contaminante a 37 °C por 1 h.
A biblioteca de microssatélites nucleares, obtida por meio de sequenciamento shotgun do DNA genômico de P. solimoesense em um sequenciador Roche 454 GSFLX (Roche Diagnostics) como descrito por Wörmann et al. (2012), foi utilizada para a mineração e a caracterização das regiões de SSR no presente estudo. A ocorrência e as frequências dos SSRs foram determinadas utilizando-se o software SciRoKo (Kofler et al., 2007) e os primers para a amplificação de tais marcadores foram desenhados a partir da ferramenta WebSat (Martins et al., 2009). Adicionalmente, as características termodinâmicas e de interação entre os pares de primers obtidos foram testados no software PerlPrimer (Marshall, 2004).
RESULTADOS E DISCUSSÃO
Nesta primeira etapa do projeto, foram utilizados espécimes de diferentes populações de Philodendron oriundas da Amazônia e da Floresta Atlântica. As folhas jovens destes indivíduos estavam devidamente armazenadas nas dependências do Laboratório de Genética e Biotecnologia Vegetal (LGBV) do Departamento de Genética da Universidade Federal de Pernanbuco (UFPE). O número de espécimes utilizados, até o momento, e outras informações dessa etapa podem ser visualizadas na Tabela 1.

No presente momento, está sendo realizada a extração de DNA de espécimes de populações adicionais das espécies já amostradas, bem com todo o processo de purificação do DNA.
A partir dos dados obtidos por meio do sequenciamento shotgun do DNA genômico de P. solimoesense foram sequenciados 46.321 fragmentos, dos quais 1.168 contêm motivos SSR aptos a serem utilizados no desenho e posterior síntese dos 20 pares de primers a serem empregados nas análises deste trabalho. Em relação à composição de microssatélites, um total de 2.299 loci de SSR foram encontrados (Tabela 2). Repetições de dinucleotídeos foram mais frequentes do que os outros tipos de repetições, apresentando 953 SSRs (41,5%), das quais 631 (66,2%) foram motivos AG/CT. Em contrapartida, foram contadas apenas 115 repetições de mononucleotídeos (5%), das quais a grande maioria (88,7%) consistiu em SSRs poli-A. Com base nesses dados, observa-se que aproximadamente 20% das regiões aptas para serem utilizadas no desenho de primers continham repetições SSRs, com no mínimo 20 pb, que podem ser devidamente testadas para a amplificação de loci polimórficos e utilizadas em mais análises de diversidade populacional em espécies de P. solimoesense, como também para análises de amplificação cruzada em outras espécies do gênero (ver Vasconcelos et al., 2013).
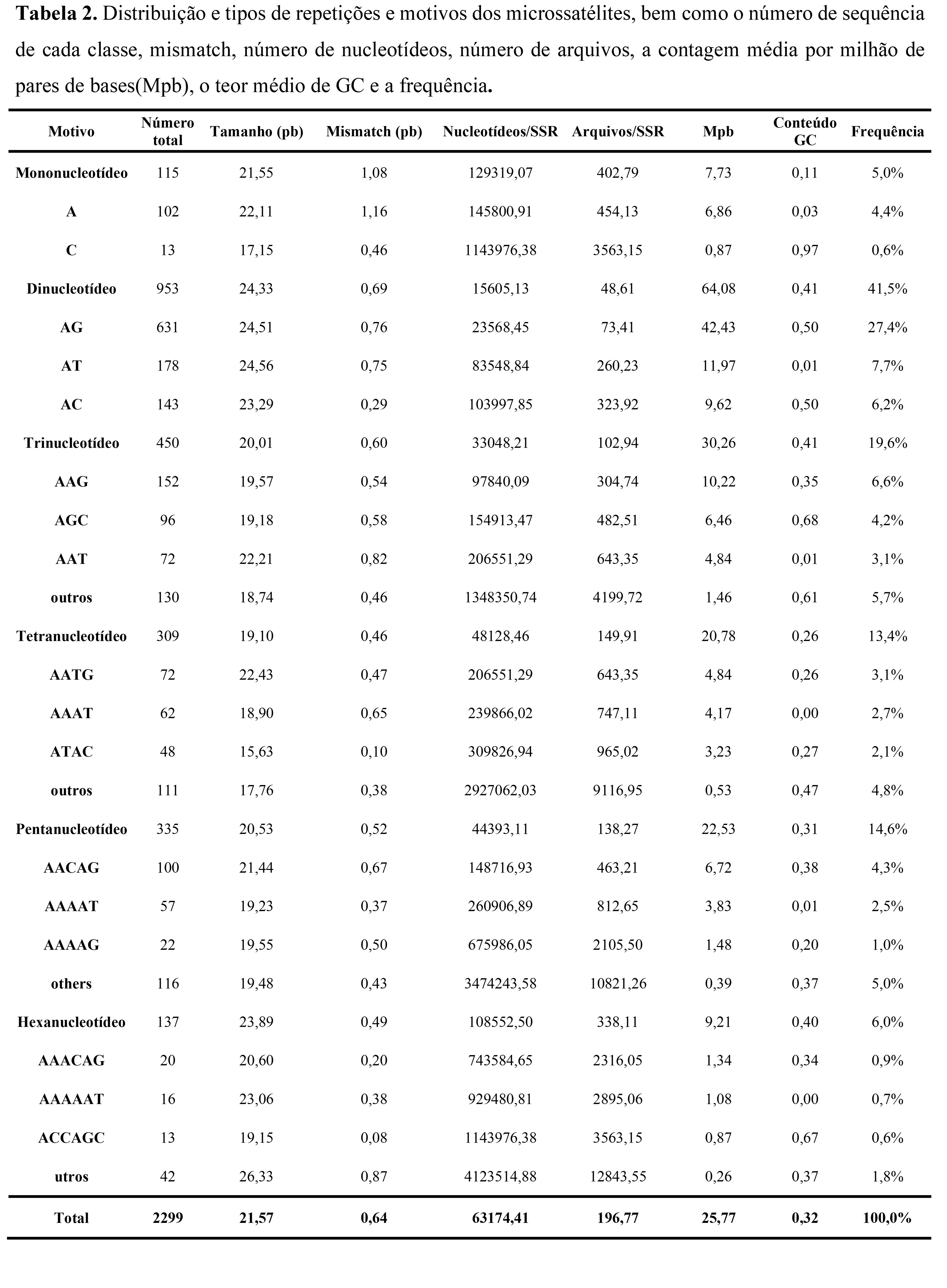
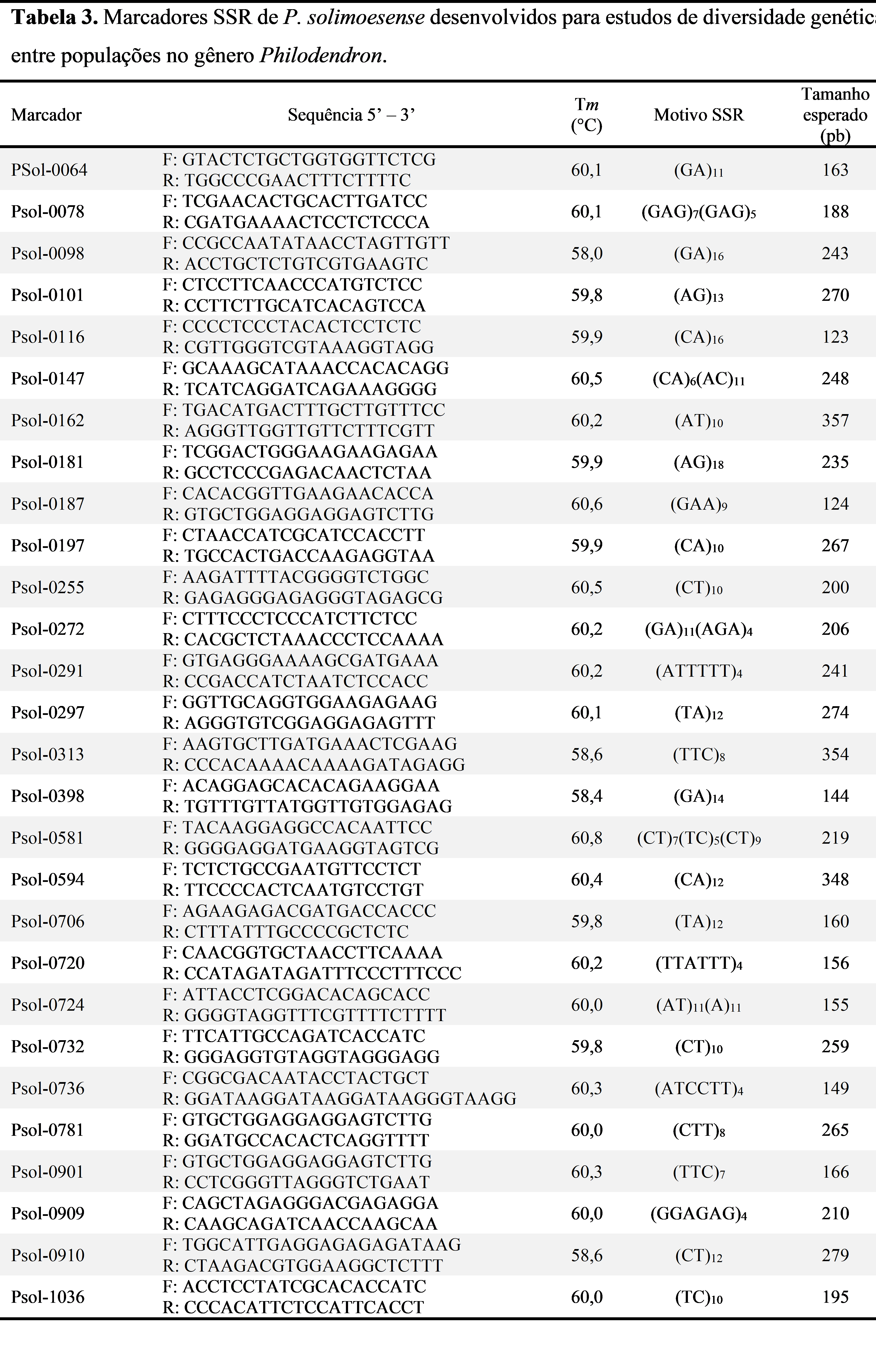
Um total de 1.168 regiões foram apropriadas para o desenho de marcadores SSR, constituindo mais de 50% de todas as regiões observadas. Destas, as 28 regiões contendo os maiores números de repetições foram selecionadas para a confecção dos respectivos primers (Tabela 3), os quais já se encontram no LGBV - UFPE.
CONCLUSÃO
Os pares de primers SSR avaliados possuem grande potencial para estudos genéticos no gênero Philodendron, tais como estudos de variabilidade intraespecíficos e interespecíficos. Dessa forma, os fragmentos amplificados serão separados em gel de poliacrilamida (6%), pois o poder de resolução é maior para análises de polimorfismos. Além disso, novos ensaios envolvendo espécimes de diferentes populações de Philodendron, oriundas de quatro regiões da Amazônia e de duas regiões da Floresta Atlântica estão sendo utilizados, totalizando 68 indivíduos. Considerando-se o alto custo no desenvolvimento de marcadores SSR, a análise de transferibilidade mostrou-se relevante e fundamental para a identificação da diversidade genética em populações de Philodendron.
REFERÊNCIAS
BATALHA-FILHO, H., FJELDSÅ, J., FABRE, P. H., & MIYAKI, C. Y. (2013) Connections between the Atlantic and the Amazonian forest avifaunas represent distinct historical events. Journal of Ornithology, v. 54, p. 41-50.
CASTELLAR, A.; OLIVEIRA, D. R.; LEITÃO, S. G.; BIZZO, H. R.; SOARES, M. D. L. C.; KINUPP, V. F.; VEIGA-JUNIOR, V. F. (2013) Essential oil from Philodendron fragrantissimum, an aromatic Araceae from Amazonia, Brazil. Journal of Essential Oil Research, v. 25, p. 194-197.
COSTA, L. P. (2003) The historical bridge between the Amazon and the Atlantic Forest of Brazil: a study of molecular phylogeography with small mammals. Journal of Biogeography, v. 30, p. 71-86.
CROAT, T. (1997) A revision of Philodendron subgenus Philodendron (Araceae) for Mexico and Central America. Annals of the Missouri Botanical Garden, v. 84, p. 311-704, 1997.
GAUTHIER, M. P. L.; BARABE, D.; BRUNEAU, A. (2008) Molecular phylogeny of the genus Philodendron (Araceae): delimitation and infrageneric classification. Botanical Journal of the Linnean Society, v. 156, p. 13-27.
GRAYMUM, M. H. (1996) Revision of Philodendron subgenus Pteromischum (Araceae) for Pacific and Caribbean Tropical America. Systematic Botany Monographs, v. 47, p, 1-233.
KOFLER, R.; SCHLOTTERER, C.; LELLEY, T. (2007) SciRoKo: a new tool for whole genome microsatellite search and investigation. Bioinformatics, v. 23, p. 1683-1685.
MARSHALL, O. J. (2004) PerlPrimer: cross-platform, graphical primer design for standard, bisulphite and real-time PCR. Bioinformatics, v. 20, p. 2471-2472.
MARTINS, W. S.; LUCAS, D. C. S.; DE SOUZA NEVES, K. F.; BERTIOLI, D. J. (2009) WebSat - a web software for microsatellite marker development. Bioinformation, v. 3, n. 6, p. 282-283.
MAYO, S. J. (1991) A revision of Philodendron subgenus Meconostigma (Araceae). Kew Bulletin, v. 46, p. 601-681, 1991.
MAYO, S. J.; BOGNER, J.; BOYCE, P. C. (1997) The genera of Araceae. Royal Botanic Gardens, Kew, Richmond, Surrey, UK. 346p.
MIYAKI, C. Y.; ALVES, M. A. S. (2006) Técnicas genéticas aplicadas à conservação, In: ROCHA, C. F. D. et al. (Orgs). Biologia da Conservação: essências. São Carlos: RiMa. p. 437-458.
OLIVEIRA, R. F. M. (2011) Aspectos etnobotânicos e taxonômicos de Araceae Juss. na comunidade Santa Maria, Baixo Rio Negro - AM. Dissertação (Mestrado em Ciências Biológicas - Botânica) - Instituto Nacional de Pesquisas da Amazônia, Manaus.
PARK, S. D. E. (2001) Trypanotolerance in West African cattle and the population genetic effects of selection. Tese (Ph. D.) - Universidade de Dublin, Dublin, Irlanda.
PENNINGTON, R. T.; DICK, C. W. (2010) Diversification of the Amazonian flora and its relation to key geological and environmental events: a molecular perspective, In: HOORN, C.; WESSELINGH, F. P. (Orgs). Amazonia, landscape and species evolution: a look into the past. Hoboken: Wiley-Blackwell Publishing Ltd. p. 373-385.
THE PLANT LIST. Disponível em: <http://www.theplantlist.org/>. Acesso em: 10 jan. 2023.
TURCHETTO-ZOLET, A. C.; CRUZ, F.; VENDRAMIN, G. G.; SIMON, M. F.; SALGUEIRO, F.; MARGIS-PINHEIRO, M.; MARGIS, R. (2012) Large-scale phylogeography of the disjunct Neotropical tree species Schizolobium parahyba (Fabaceae - Caesalpinioideae). Molecular and Phylogenetics and Evolution, v. 65, p. 174-182.
VASCONCELOS, S. et al. (2013) Characterization of microsatellites and gene content from genome shotgun sequences of Philodendron solimoesense (Araceae), a thermogenic arum lily from the Amazon basin. Anais do IV Simpósio Brasileiro de Genética Molecular de Plantas.
VICTORIA, F. C.; DA MAIA, L. C.; DE OLIVEIRA, A. C. (2011) In silico comparative analysis of SSR markers in plants. BMC Plant Biology, v. 11, p. 15.
WEISING, K.; NYBOM, H.; PFENNINGER, M.; WOLFF, K.; KAHL, G. (2005) DNA fingerprinting in plants: principles, methods, and applications. 2ª ed. - CRC Press: Boca Raton.
WÖHRMANN, T.; WAGNER, N.; KRAPP, F.; HUETTEL, B.; WEISING, K. (2012) Development of microsatellite markers in Fosterella rusbyi (Bromeliaceae) using 454 pyrosequencing. American Journal of Botany, v. 94, n. 4
_______________________________________________
*Professor de Ciências Naturais (CMR), Doutor Biotecnologia (UFPE). Email:

